Ток станок 1м61: 1М61 Станок токарно-винторезный универсальный. Паспорт, схемы, характеристики, описание
Содержание
Виды станков
В зависимости от основных операций обработки станки с ЧПУ объединены в различные технологические группы.
Токарные станки с ЧПУ являются наиболее многочисленной группой в парке станков с ЧПУ. Их выпускают в следующих исполнениях: центровые, патронные, патронно-центровые и карусельные. В основном токарные станки имеют горизонтально расположенную ось шпинделя. Исключение составляют двухсуппортные станки и карусельные станки для обработки крупных деталей. По расположения направляющих суппорта токарные станки с ЧПУ выпускают с горизонтальным, вертикальным или наклонным расположением. Станки с вертикальными и наклонными направляющими оригинальны в своем исполнении и имеют следующие преимущества: удобство обслуживания, облегчение схода и удаление стружки, расположение ходового винта станка между направляющими, что способствует повышению точности перемещения суппорта.
Расточные станки с ЧПУ можно разделить на две основные группы: с горизонтальным или вертикальным расположением шпинделя. На расточных станках фрезеруют плоскости и пазы, сверлят и зенкеруют отверстия, растачивают отверстия, подрезают торцы, нарезают резьбу метчиками. На расточных станках с вертикальным расположением шпинделя целесообразно обрабатывать плоские заготовки, на горизонтально-расточных – корпусные детали.
На расточных станках фрезеруют плоскости и пазы, сверлят и зенкеруют отверстия, растачивают отверстия, подрезают торцы, нарезают резьбу метчиками. На расточных станках с вертикальным расположением шпинделя целесообразно обрабатывать плоские заготовки, на горизонтально-расточных – корпусные детали.
Сверлильные станки с ЧПУ изготавливают в двух исполнениях: вертикально-сверлильные и радиально-сверлильные. На них можно выполнять разнообразные работы: сверление, зенкование, зенкерование, развертывание, нарезание резьб, фрезерование и т.д. Наличие крестового стола, возможность работать последовательно несколькими инструментами, а в некоторых случаях и многоинструментальными головками значительно расширяют возможности станка.
Фрезерные станки с ЧПУ компонуют по типу вертикальных и горизонтальных консольных и бесконсольных одно- и двухжстоечных станков. Горизонтально-фрезерные станки оснащают поворотным столом, управляемым по программе. На фрезерных станках с вертикальным шпинделем преимущественно изготавливают плоскостные и коробчатой формы детали небольших габаритных размеров, а также сложные поверхности плоских и объемных кулачков, шаблонов и других деталей. На станках с горизонтальным шпинделем обрабатывают поверхности корпусных деталей, расположенные в различных плоскостях.
На станках с горизонтальным шпинделем обрабатывают поверхности корпусных деталей, расположенные в различных плоскостях.
Многоцелевые станки обеспечивают выполнение большой номенклатуры технологических операций без перебазирования детали и с автоматической сменой инструмента. Режущий инструмент расположен в специальных инструментальных магазинах большой емкости, что дает возможность в соответствии с принятой программой автоматически устанавливать в шпинделе станка любой инструмент, требуемый для обработки соответствующей поверхности детали.
Многоцелевые станки отличаются особо высокой концентрацией обработки. На них производят черновую, получистовую и чистовую обработку сложных корпусных заготовок, содержащих десятки обрабатываемых поверхностей, выполняют самые разнообразные технологические переходы: фрезерование плоскостей, уступов, канавок, окон, колодцев; сверление, зенкерование, развертывание, растачивание гладких и ступенчатых отверстий; растачивание отверстий инструмента с тонким регулированием на размер: обработку наружных и внутренних поверхностей и др.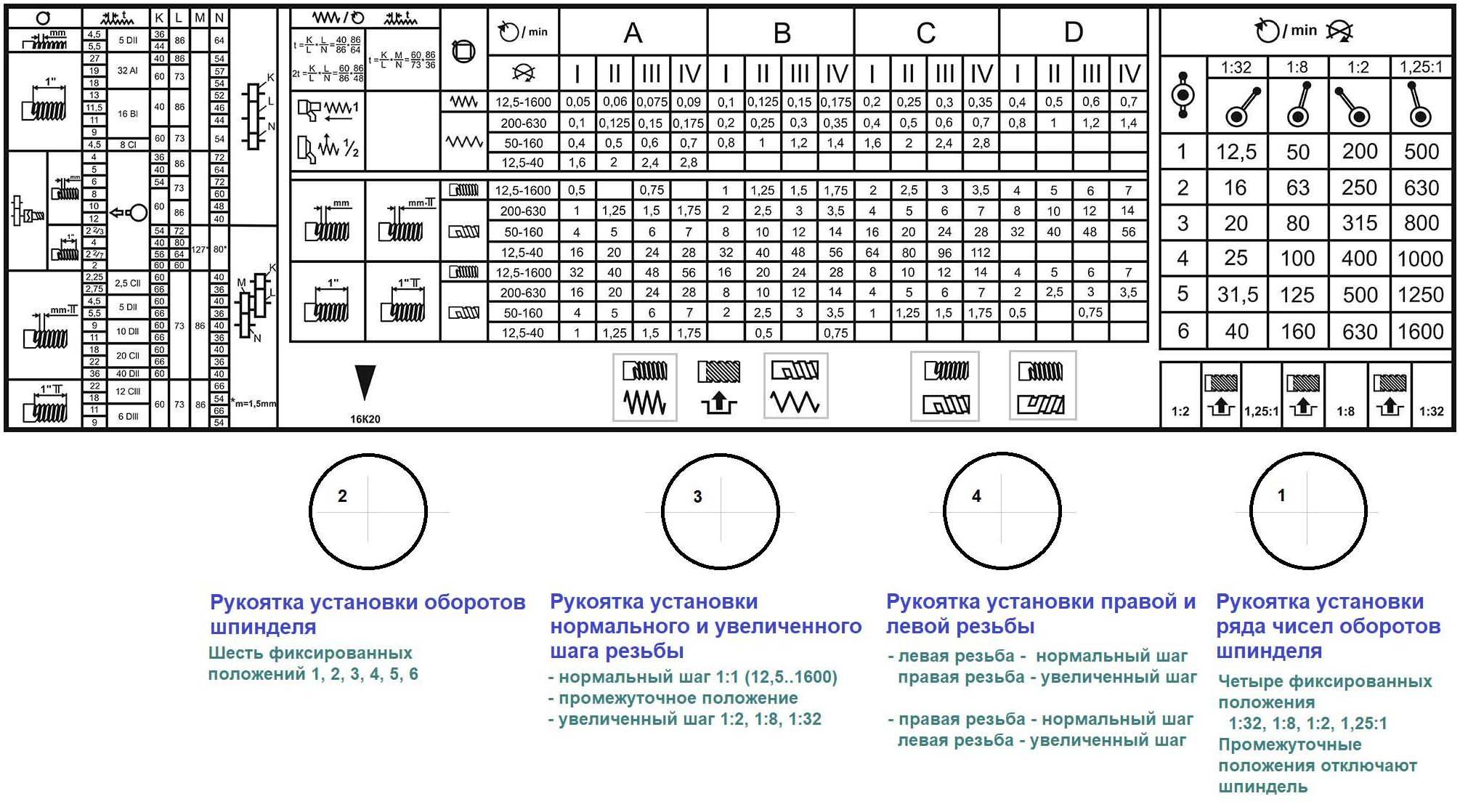
Примеры станков
ТОКАРНЫЕ СТАНКИ: МК6056, КЖ1962, РТ401, РТ403, ТСА-160, CN50A(TOS), ТС-70, 1К341, ИЖ1ИС611В, ИЖ250ИТПМ , УТ16ПМ, 16И05АФ10, 16К40 , 16В20, 1М61, 16Б16, 1А616, 1П365, 1Г340П, ТВА, 1М63, 1М63Б, 1М63Ф101, 1М63БФ101, 1К62, 1К62Д, 1К625, 1К625Д, 16К20, 16К20М, 16К20 с ЧПУ, 16К25, 1М65, 165, 1510, 1531М, 1541, 1553, 1Л532, 1М557, 1525.
ТОКАРНЫЕ АВТОМАТЫ И ПОЛУАВТОМАТЫ: 1И125П, 1И140П, 1П752МФ305, 1Д112, SARO25B, 1В116П, AWA-10M, АТС45, 1А240П-6, 1Б240-6, 1Б240-6К, 1Б265НП-6К, 1К282, 1Е365БП.
ФРЕЗЕРНЫЕ СТАНКИ: 7216Г, 65А60Ф131, 6А56, 6650, 692Р-1, 6Р13, 6Р13Ф3, 6Т12-1, 6М13СН2, FU400 (Heckert), 6Р81, 6Р81Ш, 6Р82Г, СФ-676, 6Т82-1, ВМ127, СФ35, УФ, 5К328А, 53А30П, ЕЗС380.31(аналог 53А11), 6Г463.
ШЛИФОВАЛЬНЫЕ СТАНКИ: 3М131, 3У131, COBURG, 3А183, 3Б722, 3Д711АФ11, 3Л722А, 3Е711В, 3Б70В, 3Г71, 3Г71М, SPC20b, 3У10А, HAUSER 3SM, 3Д180, 3Б633, 3К634, 3В853.
РАСТОЧНЫЕ СТАНКИ: 2Н636, 2Н636ГФ3, 2Б635, 2Д450П, 2А614-1, 2А622-1, 2А470, 2Е450АМФ4, 2254ВМФ4, 2620, 2620Г, 2620ГФ1
СВЕРЛИЛЬНЫЕ СТАНКИ: 2Н118, 2Н125, 2С132, 2Н135, 2532Л, 2А554, 2А554Ф1, 2М55.
ДОЛБЁЖНЫЕ СТАНКИ: 5А140П, 7Д430,
ОТРЕЗНЫЕ СТАНКИ: 8В66А, 8Г662, ВТС-50
ЗАТОЧНЫЕ СТАНКИ: 3Д642, 3Д692, 3Б662ВФ2, NUA-25M (аналог 3Е642, 3Е642Е), ТчПА-7.
ТРУБОНАРЕЗНЫЕ СТАНКИ: 9М14, 1Н983
ПРОЧИЕ СТАНКИ: 3Е816Ф1, 4Л723, 5С276П, Sunnen 1804, Agiecut 200, 4732ФЗМ, 5А714, HS-300.125, Машина плазменной резки.
Установка резца на токарном станке
Разберемся, зачем производить установку резца в соответствии с определенными правилами, а также рассмотрим эти правила и некоторые другие нюансы подробнее.
Вся работа на токарном оборудовании: сам процесс обработки заготовки и итоговый результат, напрямую зависят от правильности установки рабочей оснастки в держателе резца. Если резец зафиксирован неверно, то происходит быстрый износ его режущей кромки, возможны повреждения заготовки, а также возникновение нештатных ситуаций (вылет и срыв резца), которые могут навредить здоровью мастера. Кроме того может быть поврежден сам токарный станок, а его ремонт мало того стоит недешево, так еще и негативно отражается на производительности предприятия, где установлено оборудование.
До монтажа резца в держатель требуется тщательно очистить его опорные поверхности от пыли, стружки и других накопившихся загрязнений.
Основное правило расположения резца при работе на токарном станке: вершина оснастки обязана располагаться на одной линии с центральными осями зажимов токарного станка.
Если работать резцом, установленным ниже данной линии, то деталь будет выталкиваться из центров при набегании оснастки, в случае слишком высокого расположения резца он будет слишком интенсивно изнашиваться и нагреваться в процессе работы. Допустимы только небольшие отклонения от центральной линии, если этого требует конкретная операция обработки заготовки. Например, при предварительной обработке заготовки оснастку немного задирают (отклонение до 1,2 мм), а при чистовой обработке наоборот можно занижать резец на аналогичную величину.
После фиксации резца обоими болтами в держателе для регулировки по высоте необходимо подвести вплотную к центру задней или передней бабки, причем сама настройка производится при помощи подкладок, количество которых не должно превышать трех штук. Такая методика позволяет добиться максимальных показателей точности и жесткости установки резца относительно обрабатываемой детали.
Такая методика позволяет добиться максимальных показателей точности и жесткости установки резца относительно обрабатываемой детали.
Подкладки должны быть заранее заготовлены и разложены по размерам (их размер должен совпадать по сечению с площадью контакта держателя и резца), не рекомендуется в спешке применять вместо них куски различных материалов. Располагать подкладки необходимо на опорной площадке держателя оснастки, не допуская слишком большого вылета резца, который не должен быть больше высоты стержня более, чем в полтора раза, так как это может повлечь возникновение вибрации детали при ее обработке.
По глубине резец настраивается двумя способами:
- методика пробных стружек, в процессе которой оснастку приближают до касания к заготовке на включенном станке, а затем отводят до упора вправо и начинают работу, регулируя глубину на глаз;
- способ с лимбом поперечной подачи заключается в использовании встроенного средства измерения и его шкалы.

PON-SC – программа для выявления стерических столкновений, вызванных аминокислотными заменами
1. Вихинен М. Типы и эффекты белковых вариаций. Хам Жене. 2015; 134:405–421. doi: 10.1007/s00439-015-1529-6. [PubMed] [CrossRef] [Google Scholar]
2. Нироула А., Уролагин С., Вихинен М. PON-P2: метод прогнозирования для быстрой и надежной идентификации вредных вариантов. ПЛОС Один. 2015;10(2):e0117380. doi: 10.1371/journal.pone.0117380. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
3. Кирхер М., Виттен Д.М., Джейн П., О’Роак Б.Дж., Купер Г.М. Общая основа для оценки относительной патогенности генетических вариантов человека. Нат Жене. 2014;46:310–315. doi: 10.1038/ng.2892. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
4. Schwarz JM, Cooper DN, Schuelke M, Seelow D. MutationTaster2: предсказание мутаций для возраста глубокого секвенирования. Нат Методы. 2014; 11: 361–362. doi: 10.1038/nmeth.2890. [PubMed] [CrossRef] [Google Scholar]
5. Yang Y, Chen B, Tan G, Vihinen M, Shen B. Структурное предсказание влияния миссенс-варианта на стабильность белка. Аминокислоты. 2013;44:847–855. doi: 10.1007/s00726-012-1407-7. [PubMed] [CrossRef] [Академия Google]
Yang Y, Chen B, Tan G, Vihinen M, Shen B. Структурное предсказание влияния миссенс-варианта на стабильность белка. Аминокислоты. 2013;44:847–855. doi: 10.1007/s00726-012-1407-7. [PubMed] [CrossRef] [Академия Google]
6. Ченг Дж., Рэндалл А., Балди П. Прогнозирование изменений стабильности белка для односайтовых мутаций с использованием машин для опорных векторов. Белки. 2006;62:1125–1132. doi: 10.1002/прот.20810. [PubMed] [CrossRef] [Google Scholar]
7. Инь С., Дин Ф., Дохолян Н.В. Eris: автоматизированный анализатор стабильности белков. Нат Методы. 2007; 4: 466–467. doi: 10.1038/nmeth0607-466. [PubMed] [CrossRef] [Google Scholar]
8. Али Х., Уролагин С., Гурарслан О., Вихинен М. Эффективность программ прогнозирования белковых нарушений по аминокислотным заменам. Хум Мутат. 2014;35:794–804. doi: 10.1002/humu.22564. [PubMed] [CrossRef] [Google Scholar]
9. Conchillo-Sole O, de Groot NS, Aviles FX, Vendrell J, Daura X, Ventura S. AGGRESCAN: сервер для прогнозирования и оценки «горячих точек» агрегация полипептидов. Биоинформатика BMC. 2007; 8:65. дои: 10.1186/1471-2105-8-65. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Биоинформатика BMC. 2007; 8:65. дои: 10.1186/1471-2105-8-65. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
10. Trovato A, Seno F, Tosatto SC. Сервер PASTA для прогнозирования агрегации белков. Белок Eng Des Sel. 2007; 20: 521–523. дои: 10.1093/белок/gzm042. [PubMed] [CrossRef] [Google Scholar]
11. Лаурила К., Вихинен М. PROlocalizer: интегрированный веб-сервис для предсказания субклеточной локализации белка. Аминокислоты. 2011;40:975–980. doi: 10.1007/s00726-010-0724-y. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
12. Desmet FO, Hamroun D, Lalande M, Collod-Beroud G, Claustres M, Beroud C. Human Splicing Finder: онлайн-инструмент биоинформатики для прогнозирования сплайсинга сигналы. Нуклеиновые Кислоты Res. 2009;37(9):e67. дои: 10.1093/нар/гкп215. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
13. Морт М., Стерн-Вейлер Т., Ли Б., Болл Э.В., Купер Д.Н., Радивояк П., Сэнфорд Дж.Р., Муни С.Д. Сращивание MutPred: основанное на машинном обучении предсказание экзонных вариантов, которые нарушают сплайсинг. Геном биол. 2014;15:Р19. doi: 10.1186/gb-2014-15-1-r19. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Геном биол. 2014;15:Р19. doi: 10.1186/gb-2014-15-1-r19. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
14. Niroula A, Vihinen M. PON-мт-тРНК: многофакторный вероятностный метод классификации вариаций митохондриальной тРНК. Нуклеиновые Кислоты Res. 2016;44:2020–2027. дои: 10.1093/нар/gkw046. [бесплатная статья ЧВК] [PubMed] [CrossRef] [Google Scholar]
15. Кондрашов Ф.А. Предсказание патогенных мутаций в митохондриально кодируемых тРНК человека. Хум Мол Жене. 2005;14:2415–2419. doi: 10.1093/hmg/ddi243. [PubMed] [CrossRef] [Google Scholar]
16. Нироула А., Вихинен М. Предикторы интерпретации вариаций: принципы, типы, производительность и выбор. Хум Мутат. 2016; 37: 579–597. doi: 10.1002/humu.22987. [PubMed] [CrossRef] [Google Scholar]
17. Тусберг Дж., Вихинен М. Патогенны или нет? И если да, то как? Изучение эффектов миссенс-мутаций методами биоинформатики. Хум Мутат. 2009 г.;30:703–714. doi: 10.1002/humu.20938. [PubMed] [CrossRef] [Google Scholar]
18. Lindor NM, Guidugli L, Wang X, Vallee MP, Monteiro AN, Tavtigian S, Goldgar DE, Couch FJA. Обзор многофакторной вероятностной модели для классификации вариантов BRCA1 и BRCA2 с неопределенной значимостью (VUS) Hum Mutat. 2012;33:8–21. doi: 10.1002/humu.21627. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Lindor NM, Guidugli L, Wang X, Vallee MP, Monteiro AN, Tavtigian S, Goldgar DE, Couch FJA. Обзор многофакторной вероятностной модели для классификации вариантов BRCA1 и BRCA2 с неопределенной значимостью (VUS) Hum Mutat. 2012;33:8–21. doi: 10.1002/humu.21627. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
19. Goldgar DE, Easton DF, Byrnes GB, Spurdle AB, Iversen ES, Greenblatt MS. Генетические данные и интеграция различных источников данных для классификации сомнительных вариантов в единую модель. Хум Мутат. 2008;29: 1265–1272. doi: 10.1002/humu.20897. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
20. Niroula A, Vihinen M. Классификация аминокислотных замен в белках репарации несоответствия с использованием PON-MMR2. Хум Мутат. 2015; 36:1128–1134. doi: 10.1002/humu.22900. [PubMed] [CrossRef] [Google Scholar]
21. Chao EC, Velasquez JL, Witherspoon MS, Rozek LS, Peel D, Ng P, Gruber SB, Watson P, Rennert G, Anton-Culver H, et al. Точная классификация миссенс-вариантов MLh2/MSh3 с помощью многомерного анализа белковых полиморфизмов-восстановления несоответствия (MAPP-MMR) Hum Mutat. 2008;29: 852–860. doi: 10.1002/humu.20735. [PubMed] [CrossRef] [Google Scholar]
2008;29: 852–860. doi: 10.1002/humu.20735. [PubMed] [CrossRef] [Google Scholar]
22. Валиахо Дж., Фейсал И., Ортутай С., Смит CIE, Вихинен М. Характеристика всех возможных замен аминокислот, вызванных изменением одного нуклеотида, в киназном домене тирозинкиназы Брутона. Хум Мутат. 2015; 36: 638–47. doi: 10.1002/humu.22791. [PubMed] [CrossRef] [Google Scholar]
23. Niroula A, Vihinen M. Прогнозирование тяжести болезнетворных вариантов. Хум Мутат. 2017; 38: 357–364. doi: 10.1002/humu.23173. [PubMed] [CrossRef] [Академия Google]
24. Шаповалов М.В., Данбрэк Р.Л. Младший. Сглаженная зависимая от остова библиотека ротамеров для белков, полученная на основе адаптивных оценок плотности ядер и регрессий. Структура (Лондон, Англия). 2011;19:844–58. [Бесплатная статья PMC] [PubMed]
25. Lovell SC, Word JM, Richardson JS, Richardson DC. Предпоследняя библиотека ротамеров. Белки. 2000;40:389–408. doi: 10.1002/1097-0134(20000815)40:3<389::AID-PROT50>3.0.CO;2-2. [PubMed] [CrossRef] [Google Scholar]
26. Towse CL, Rysavy SJ, Vulovic IM, Daggett V. Новые динамические библиотеки ротамеров: управляемый данными анализ конформационных склонностей боковых цепей. Структура (Лондон, Англия). 2016; 24: 187–99. [Статья бесплатно PMC] [PubMed]
Towse CL, Rysavy SJ, Vulovic IM, Daggett V. Новые динамические библиотеки ротамеров: управляемый данными анализ конформационных склонностей боковых цепей. Структура (Лондон, Англия). 2016; 24: 187–99. [Статья бесплатно PMC] [PubMed]
27. Кривов Г.Г., Шаповалов М.В., Данбрэк Р.Л., мл. Улучшенное предсказание конформаций боковых цепей белков с помощью SCWRL4. Белки. 2009; 77: 778–795. doi: 10.1002/прот.22488. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
28. Нагата К., Рэндалл А., Балди П. SIDEpro: новый подход к машинному обучению для быстрого и точного прогнозирования конформаций боковой цепи. Белки. 2012; 80: 142–153. doi: 10.1002/прот.23170. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
29. Райт Дж. Д., Лим, Калифорния. Быстрый метод предсказания аминокислотных мутаций, ведущих к разворачиванию. Белок англ. 2001; 14: 479–486. doi: 10.1093/белок/14.7.479. [PubMed] [CrossRef] [Google Scholar]
30. Word JM, Bateman RC, Jr, Presley BK, Lovell SC, Richardson DC. Изучение стерических ограничений белковых мутаций с помощью MAGE/PROBE. Белковая наука. 2000;9:2251–9. doi: 10.1110/ps.9.11.2251. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Изучение стерических ограничений белковых мутаций с помощью MAGE/PROBE. Белковая наука. 2000;9:2251–9. doi: 10.1110/ps.9.11.2251. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
31. Pottel J, Moitessier N. Одноточечная мутация с помощью набора инструментов библиотеки Rotamer: к белковой инженерии. Модель J Chem Inf. 2015;55:2657–2671. doi: 10.1021/acs.jcim.5b00525. [PubMed] [CrossRef] [Академия Google]
32. Rouet R, Lowe D, Christ D. Разработка стабильности репертуара человеческих антител. ФЭБС лат. 2014; 588: 269–277. doi: 10.1016/j.febslet.2013.11.029. [PubMed] [CrossRef] [Google Scholar]
33. Соча Р.Д., Токурики Н. Модулирование стабильности белка – стратегии направленной эволюции для улучшения функции белка. FEBS J. 2013; 280: 5582–5595. doi: 10.1111/февраль 12354. [PubMed] [CrossRef] [Google Scholar]
34. O’Fagain C. Инженерия стабильности белков. Методы молекулярной биологии (Клифтон, Нью-Джерси) 2011;681:103–136. дои: 10.1007/978-1-60761-913-0_7. [PubMed] [CrossRef] [Google Scholar]
[PubMed] [CrossRef] [Google Scholar]
35. Poultney CS, Butterfoss GL, Gutwein MR, Drew K, Gresham D, Gunsalus KC, Shasha DE, Bonneau R. Рациональный дизайн чувствительных к температуре аллелей с использованием предсказания вычислительной структуры. ПЛОС Один. 2011;6:e23947. doi: 10.1371/journal.pone.0023947. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
36. Tan KP, Khare S, Varadarajan R, Madhusudhan MS. TSpred: веб-сервер для рационального конструирования термочувствительных мутантов. Нуклеиновые Кислоты Res. 2014;42:W277–W284. дои: 10.1093/нар/гку319. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
37. Тусберг Дж., Вихинен М. Структурная основа гипердефицита IgM — мутации CD40L. Белок Eng Des Sel. 2007; 20: 133–141. doi: 10.1093/белок/gzm004. [PubMed] [CrossRef] [Google Scholar]
38. Тусберг Дж., Вихинен М. Биоинформатический анализ взаимосвязей структуры и функции белка: тематическое исследование миссенс-мутаций лейкоцитарной эластазы (ELA2). Хум Мутат. 2006; 27:1230–1243. doi: 10.1002/humu.20407. [PubMed] [CrossRef] [Академия Google]
Хум Мутат. 2006; 27:1230–1243. doi: 10.1002/humu.20407. [PubMed] [CrossRef] [Академия Google]
39. Cock PJ, Antao T, Chang JT, Chapman BA, Cox CJ, Dalke A, Friedberg I, Hamelryck T, Kauff F, Wilczynski B, et al. Biopython: бесплатные инструменты Python для вычислительной молекулярной биологии и биоинформатики. Биоинформатика. 2009; 25:1422–1423. doi: 10.1093/биоинформатика/btp163. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
40. Фришман Д., Аргос П. Назначение вторичной структуры белка на основе знаний. Белки. 1995; 23: 566–579. doi: 10.1002/прот.340230412. [PubMed] [CrossRef] [Академия Google]
41. Pedregosa F, Varoquaux G, Gramfort A, Michel V, Thirion B, Grisel O, Blondel M, Prettenhofer P, Weiss R, Dubourg V, et al. Scikit-learn: машинное обучение на питоне. Дж. Мах Узнать Рез. 2011;12:2825–2830. [Google Scholar]
42. Hooft RW, Vriend G, Sander C, Abola EE. Ошибки в белковых структурах. Природа. 1996; 381:272. doi: 10.1038/381272a0. [PubMed] [CrossRef] [Google Scholar]
43. Джеффри Г. Введение в водородные связи. Нью-Йорк: Издательство Оксфордского университета; 1997. [Google Scholar]
Джеффри Г. Введение в водородные связи. Нью-Йорк: Издательство Оксфордского университета; 1997. [Google Scholar]
44. Ричардсон Дж.С. Анатомия и таксономия структуры белка. Adv Protein Chem. 1981; 34: 167–339. doi: 10.1016/S0065-3233(08)60520-3. [PubMed] [CrossRef] [Google Scholar]
45. Berman HM, Westbrook J, Feng Z, Gilliland G, Bhat TN, Weissig H, Shindyalov IN, Bourne PE. Банк данных о белках. Нуклеиновые Кислоты Res. 2000; 28: 235–242. doi: 10.1093/нар/28.1.235. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
46. Nair PS, Vihinen M. VariBench: эталонная база данных для вариантов. Хум Мутат. 2013; 34:42–49. doi: 10.1002/humu.22204. [PubMed] [CrossRef] [Google Scholar]
47. Word JM, Lovell SC, LaBean TH, Taylor HC, Zalis ME, Presley BK, Richardson JS, Richardson DC. Визуализация и количественная оценка молекулярного соответствия: точки контакта небольшого зонда с явными атомами водорода. Дж Мол Биол. 1999; 285:1711–1733. doi: 10.1006/jmbi. 1998.2400. [PubMed] [CrossRef] [Google Scholar]
1998.2400. [PubMed] [CrossRef] [Google Scholar]
48. Лаппалайнен И., Тусберг Дж., Шен Б., Вихинен М. Анализ генома патогенных мутаций домена Sh3. Белки. 2008;72:779–792. doi: 10.1002/прот.21970. [PubMed] [CrossRef] [Google Scholar]
49. Ши З., Моулт Дж. Структурное и функциональное влияние миссенс-соматических мутаций, связанных с раком. Дж Мол Биол. 2011; 413:495–512. doi: 10.1016/j.jmb.2011.06.046. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
50. Вихинен М. Как оценить эффективность методов прогнозирования? Показатели и их интерпретация в анализе вариационного эффекта. Геномика ВМС. 2012;13(Приложение 4):S2. doi: 10.1186/1471-2164-13-S4-S2. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
51. Вихинен М. Руководство по отчетности и использованию инструментов прогнозирования для анализа генетической изменчивости. Хум Мутат. 2013; 34: 275–282. doi: 10.1002/humu.22253. [PubMed] [CrossRef] [Google Scholar]
52. Эяль Э., Найманович Р.
